Prima dell’avvento delle moderne tecnologie di sequenziamento del DNA negli anni duemila, solo una piccolissima percentuale di batteri appartenenti al microbiota intestinale poteva essere rilevata. Infatti, gran parte dei microrganismi appartenenti al microbiota sono definiti incoltivabili, dal momento che non possono essere isolati con le tradizionali tecniche di microbiologia classica in laboratorio.
Le moderne tecnologie NGS (Next Generation Sequencing) di sequenziamento del DNA, basate su strumenti di nuova generazione, hanno permesso di svelare l’enorme biodiversità di questa complessa comunità microbica. Questi approcci vengono definiti molecolari o coltura-indipendenti, poiché non legati alla coltivazione dei microrganismi in laboratorio.
Gli approcci coltura-indipendenti per lo studio del microbiota intestinale prevedono l’estrazione del DNA da un campione (generalmente feci o biopsia intestinale se disponibile) e il successivo sequenziamento, ovvero l’identificazione della successione delle basi puriniche e pirimidiniche nella molecola di DNA stessa.
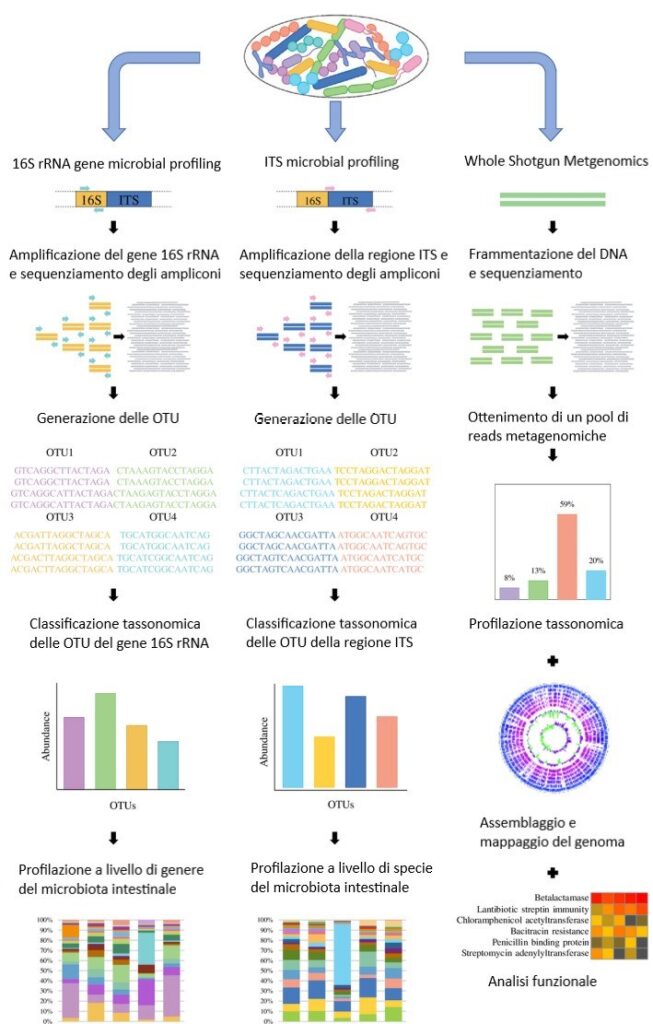
Attualmente, Attualmente, tre sono le principali metodiche sfruttate per lo studio e la caratterizzazione del microbiota intestinale: 16S rRNA gene microbial profiling, ITS microbial profiling e shotgun metagenomics.
16S rRNA gene microbial profiling
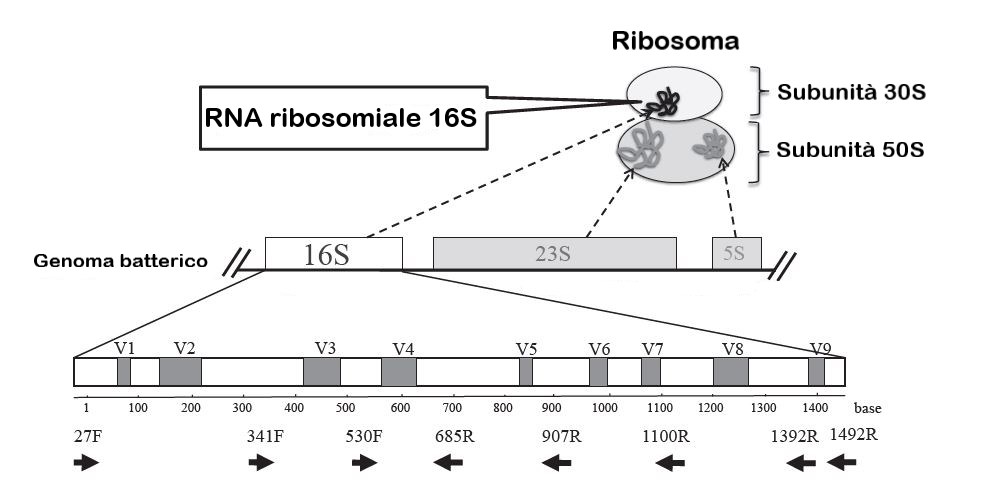
Questa metodica si basa sul sequenziamento di una piccola porzione del DNA contenuto all’interno di ogni cellula batterica, nota con il nome di gene 16S rRNA, appartenente all’operone ribosomiale e quindi presente in tutti i batteri. Questo gene è costituito da 10 regioni conservate e 6 regioni variabili. L’approccio 16S rRNA microbial profiling prevede prima l’amplificazione di una o più regioni del gene grazie all’utilizzo di primer disegnati sulle regioni conservate e il successivo sequenziamento dell’amplicone. Questa tecnica permette di discriminare microrganismi appartenenti a generi diversi, sulla base delle differenze in termini di sequenza nucleotidica a carico del gene 16S rRNA
ITS microbial profiling
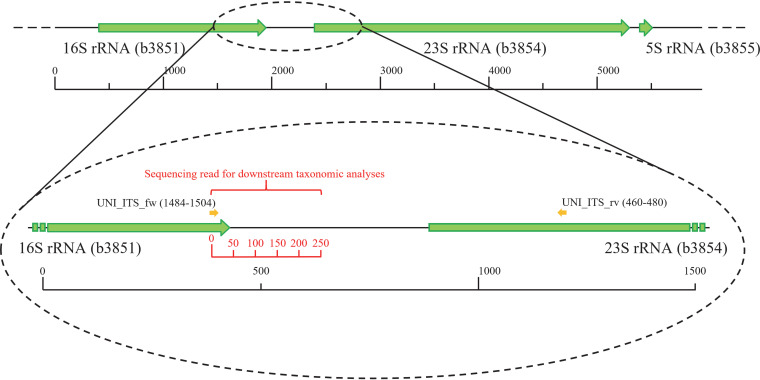
Una seconda metodologia è rappresentata dall’ITS microbial profiling, che prevede l’amplificazione della regione Internal Trascribed Spacer (ITS), collocata tra il gene 16S rRNA e 23S rRNA dell’operone ribosomiale e pertanto, come il gene 16S rRNA, anche questa regione è presente in tutti i batteri. Tale regione, però, risulta meno conservata rispetto al gene 16S rRNA. Proprio per questo, con questa tecnica è possibile ottenere una classificazione fino a livello di specie e, addirittura di sottospecie.
Shotgun metagenomics
L’approccio di shotgun metagenomics si basa, invece, sul sequenziamento massivo del DNA totale estratto da un dato campione. Questa metodologia non prevede alcuno step di amplificazione di un singolo gene o porzione di un gene. Ciò consente, non solo di ottenere una classificazione tassonomica a livello di (sotto)specie, ma anche di ricostruire i genomi dei diversi ceppi presenti all’interno del campione, consentendo a sua volta di predire le potenzialità funzionali di ciascuna specie del campione in esame. Negli ultimi anni, è stato proposto anche l’approccio shallow shotgun sequencing, ovvero un approccio shotgun metagenomics a bassa profondità che permette di ottenere una profilazione tassonomica a livello di (sotto)specie abbattendo però notevolmente i costi rispetto all’approccio shotgun metagenomics classico.
Bibliografia
- Lagier JC, Hugon P, Khelaifia S, Fournier PE, La Scola B, Raoult D: The rebirth of culture in microbiology through the example of culturomics to study human gut microbiota. Clin Microbiol Rev 2015, 28(1):237-264. https://pubmed.ncbi.nlm.nih.gov/25567229/
- Milani C, Duranti S, Bottacini F, Casey E, Turroni F, Mahony J, Belzer C, Delgado Palacio S, Arboleya Montes S, Mancabelli L et al: The First Microbial Colonizers of the Human Gut: Composition, Activities, and Health Implications of the Infant Gut Microbiota. Microbiol Mol Biol Rev 2017, 81(4). https://pubmed.ncbi.nlm.nih.gov/29118049/
- Alessandri G, Argentini C, Milani C, Turroni F, Cristina Ossiprandi M, van Sinderen D, Ventura M: Catching a glimpse of the bacterial gut community of companion animals: a canine and feline perspective. Microb Biotechnol 2020, 13(6):1708-1732. https://pubmed.ncbi.nlm.nih.gov/32864871/
- Fukuda K, Ogawa M, Taniguchi H, Saito M: Molecular Approaches to Studying Microbial Communities: Targeting the 16S Ribosomal RNA Gene. J UOEH 2016, 38(3):223-232. https://pubmed.ncbi.nlm.nih.gov/27627970/
- Milani, Christian et al. “Untangling Species-Level Composition of Complex Bacterial Communities through a Novel Metagenomic Approach.” mSystems vol. 5,4 e00404-20. 28 Jul. 2020. https://pubmed.ncbi.nlm.nih.gov/32723792/


Lascia un commento